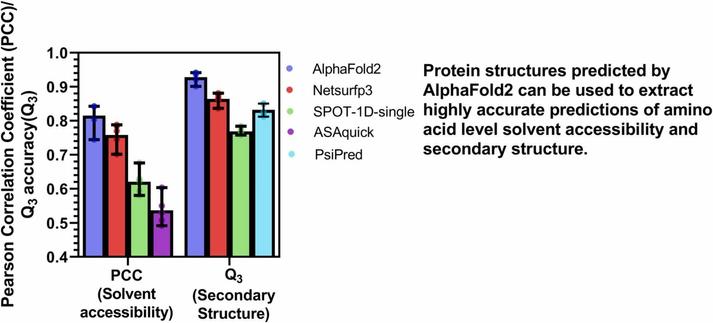
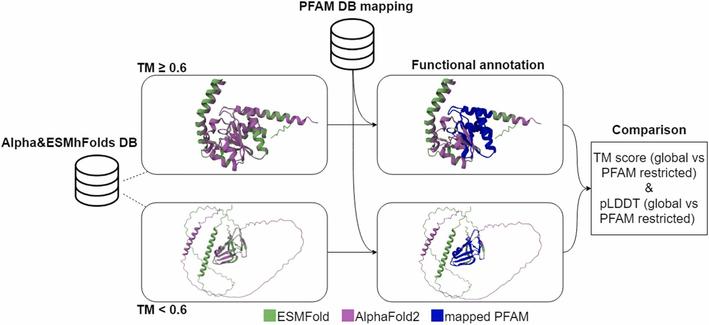
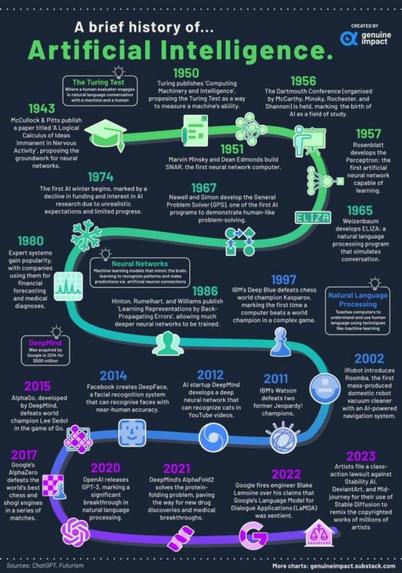
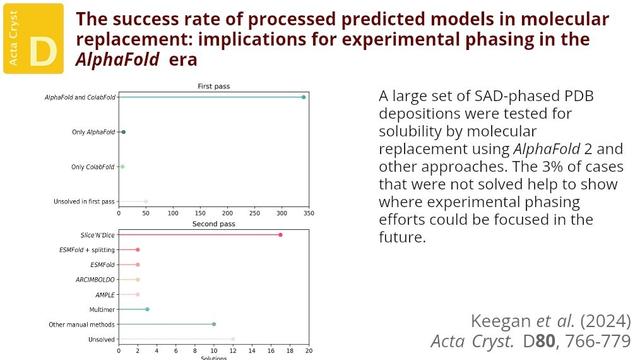
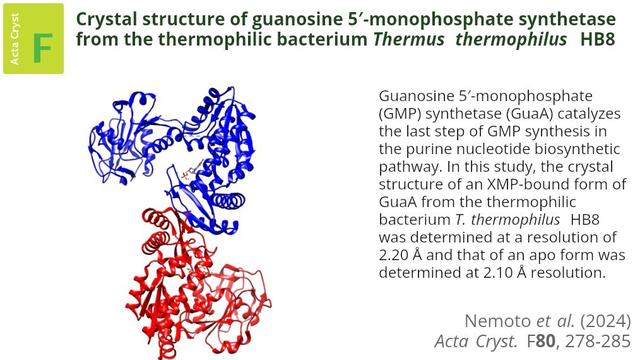
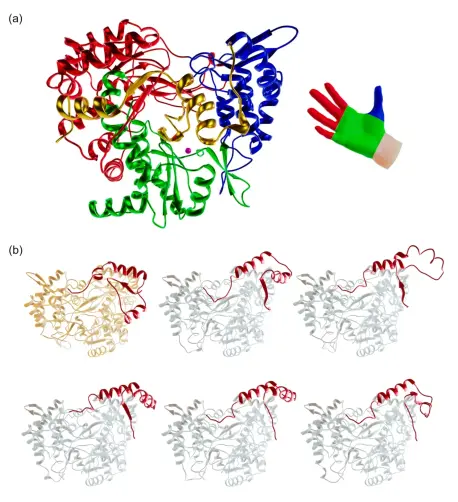
Pequeños y grandes pasos hacia el imperio de la inteligencia artificial
Fuente: Open Tech
Traducción de la infografía:
- 1943 – McCullock y Pitts publican un artículo titulado Un cálculo lógico de ideas inmanentes en la actividad nerviosa, en el que proponen las bases para las redes neuronales.
- 1950 – Turing publica Computing Machinery and Intelligence, proponiendo el Test de Turing como forma de medir la capacidad de una máquina.
- 1951 – Marvin Minsky y Dean Edmonds construyen SNAR, la primera computadora de red neuronal.
- 1956 – Se celebra la Conferencia de Dartmouth (organizada por McCarthy, Minsky, Rochester y Shannon), que marca el nacimiento de la IA como campo de estudio.
- 1957 – Rosenblatt desarrolla el Perceptrón: la primera red neuronal artificial capaz de aprender.
(!!) Test de Turing: donde un evaluador humano entabla una conversación en lenguaje natural con una máquina y un humano.
- 1965 – Weizenbaum desarrolla ELIZA: un programa de procesamiento del lenguaje natural que simula una conversación.
- 1967 – Newell y Simon desarrollan el Solucionador General de Problemas (GPS), uno de los primeros programas de IA que demuestra una capacidad de resolución de problemas similar a la humana.
- 1974 – Comienza el primer invierno de la IA, marcado por una disminución de la financiación y del interés en la investigación en IA debido a expectativas poco realistas y a un progreso limitado.
- 1980 – Los sistemas expertos ganan popularidad y las empresas los utilizan para realizar previsiones financieras y diagnósticos médicos.
- 1986 – Hinton, Rumelhart y Williams publican Aprendizaje de representaciones mediante retropropagación de errores, que permite entrenar redes neuronales mucho más profundas.
(!!) Redes neuronales: modelos de aprendizaje automático que imitan el cerebro y aprenden a reconocer patrones y hacer predicciones a través de conexiones neuronales artificiales.
- 1997 – Deep Blue de IBM derrota al campeón mundial de ajedrez Kasparov, siendo la primera vez que una computadora vence a un campeón mundial en un juego complejo.
- 2002 – iRobot presenta Roomba, el primer robot aspirador doméstico producido en serie con un sistema de navegación impulsado por IA.
- 2011 – Watson de IBM derrota a dos ex campeones de Jeopardy!.
- 2012 – La startup de inteligencia artificial DeepMind desarrolla una red neuronal profunda que puede reconocer gatos en vídeos de YouTube.
- 2014 – Facebook crea DeepFace, un sistema de reconocimiento facial que puede reconocer rostros con una precisión casi humana.
(!!) DeepMind fue adquirida por Google en 2014 por 500 millones de dólares.
- 2015 – AlphaGo, desarrollado por DeepMind, derrota al campeón mundial Lee Sedol en el juego de Go.
- 2017 – AlphaZero de Google derrota a los mejores motores de ajedrez y shogi del mundo en una serie de partidas.
- 2020 – OpenAI lanza GPT-3, lo que marca un avance significativo en el procesamiento del lenguaje natural.
(!!) Procesamiento del lenguaje natural: enseña a las computadoras a comprender y utilizar el lenguaje humano mediante técnicas como el aprendizaje automático.
- 2021 – AlphaFold2 de DeepMind resuelve el problema del plegamiento de proteínas, allanando el camino para nuevos descubrimientos de fármacos y avances médicos.
- 2022 – Google despide al ingeniero Blake Lemoine por sus afirmaciones de que el modelo de lenguaje para aplicaciones de diálogo (LaMDA) de Google era sensible.
- 2023 – Artistas presentaron una demanda colectiva contra Stability AI, DeviantArt y Mid-journey por usar Stable Diffusion para remezclar las obras protegidas por derechos de autor de millones de artistas.
Gráfico: Open Tech / Genuine Impact
Entradas relacionadas
#ajedrez #AlphaFold2 #AlphaGo #AlphaZero #aprendizajeAutomático #artículo #artistas #aspirador #BlakeLemoine #ConferenciaDeDartmouth #copyright #DeanEdmonds #DeepBlue #DeepFace #DeepMind #DeviantArt #ELIZA #Facebook #gatos #GenuineImpact #Go #Google #GPS #GPT3 #gráfico #Hinton #IA #IBM #infografía #inteligenciaArtificial #iRobot #Jeopardy_ #Kasparov #LaMDA #LeeSedol #MarvinMinsky #McCarthy #McCullock #MidJourney #modelos #Newell #OpenTech #OpenAI #patrones #Perceptron #Pitts #plegamientoDeProteínas #predicciones #procesamientoDelLenguajeNatural #reconocimientoFacial #redesNeuronales #remezclar #robot #Rochester #Roomba #Rosenblatt #Rumelhart #Shannon #shogi #Simon #sistemaDeNavegación #SNAR #StabilityAI #StableDiffusion #testDeTuring #Turing #vídeos #Watson #Weizenbaum #Williams #YouTube