[Перевод] О прионах и дизайне белков
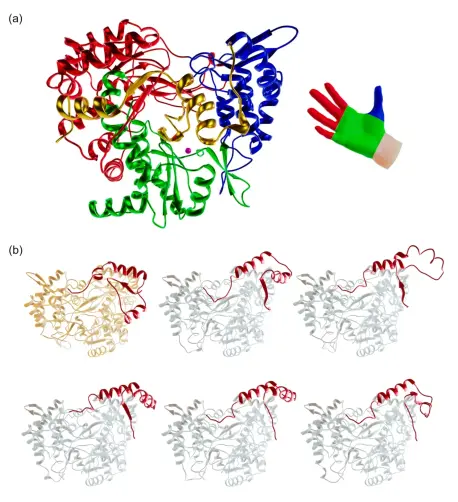
Зачастую крупные открытия в области биологии происходят при изучении крошечных существ. И многое из того, что мы сегодня считаем догмой, появилось благодаря изучению конкретных микроорганизмов. Эксперименты с бактерией Streptococcus pneumoniae не только привели к открытию ДНК как основной наследственной молекулы , но и позволили создать первые инструменты для генной инженерии. Изучение РНК опухолевых вирусов позволило обнаружить обратную транскриптазу — фермент, необходимый для изучения биологии РНК. Эти модельные микроорганизмы долгое время служили природными лабораториями, позволяя расшифровывать фундаментальные принципы жизни и предоставляя инструменты, необходимые для их изучения. Перейдя от микроорганизмов к макромолекулам, биология вступила в эру белков. Последние достижения в области искусственного интеллекта, включая такие инструменты, как нобелевский лауреат AlphaFold , сделали возможным предсказание структуры белков, превратив кропотливый лабораторный процесс в вычислительную задачу, которую можно решить за считанные минуты.
https://habr.com/ru/articles/933490/
#белки #прионы #alphafold2